Imagerie biomédicale
Le microscope optique est un outil incontournable du biologiste. Visualiser en direct des processus cellulaires aussi précis que le recrutement dynamique de calcium qui intervient dans la plasticité des neurones, ou le devenir de certaines protéines lors de la division cellulaire, sont des prouesses rendues possibles grâce au développement d'une nouvelle méthode d’imagerie appelée microscopie par localisation photo-activée (ou imagerie PALM).
Qu'est-ce que l'imagerie PALM ?
Cette méthode a obtenu le prix Nobel de Chimie en 2014. Elle permet, grâce à des techniques élaborées de traitement d’images, d’estimer la position d’une particule fluorescente avec une précision bien supérieure à la limite de diffraction et ainsi de reconstruire des images dites « super-résolues » ayant une résolution nanométrique [1]. Comme l’illustre la figure 1, l’imagerie PALM permet de révéler des détails indiscernables en imagerie conventionnelle.
Figure 1 : Image PALM (droite) de protéines bactériennes membranaires en comparaison d’une même image obtenue en microscopie conventionnelle (gauche). Crédit : Antony Lee, University of California, Berkeley.
Avec cette approche, les biologistes cherchent aujourd’hui à imager des milieux biologiques de plus en plus épais et non homogènes. Comme l’image des particules ne peut pas être parfaitement focalisée à toutes les profondeurs, cela conduit à une forte dégradation de la résolution. Il est donc nécessaire d’augmenter la « profondeur de champ » du microscope, c’est-à-dire d’améliorer la précision de localisation d’une particule le long de l’axe optique. De plus, dans certaines applications, il peut être nécessaire d’estimer la position tridimensionnelle de la particule.
Quels sont les enjeux de nos travaux de recherche ?
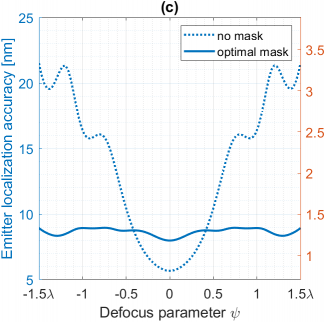
Ces défis peuvent être relevés en plaçant un masque de phase optimisé dans la pupille du microscope pour modifier sa réponse impulsionnelle lorsqu’une particule est défocalisée, et en appliquant des algorithmes de traitement adaptés. Notre équipe développe depuis plusieurs années des masques de phase placés dans la pupille d'une optique afin d'étendre sa profondeur de champ en imagerie classique [2,3]. Nous avons récemment généralisé cette approche pour développer des masques de phase et des algorithmes de traitement adaptés à la microscopie PALM [4]. Pour cela, nous mettons en œuvre une approche de « co-conception » qui consiste à optimiser les profils des masques en prenant en compte le traitement numérique appliqué aux images. D’un point de vue applicatif, nous avons montré que cette nouvelle approche augmente de manière significative la profondeur d'exploration des méthodes d'imagerie PALM (voir Figure 2), ce qui est un enjeu fondamental dans l'étude des milieux biologiques.
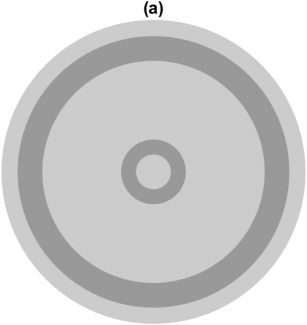
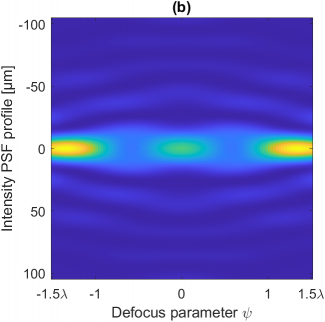
Figure 2 : (a) Masque optimal à cinq anneaux. (b) Variation du profil transversal de la PSF en fonction de la défocalisation lorsque le microscope est équipé d’un masque optimal. (c) Précision de localisation sans masque (pointillés) et avec masque (bleu) en fonction de la défocalisation.
Ces travaux sont menés en étroite collaboration avec l’équipe du laboratoire LP2N en Optique et Biologie. Cette équipe met en œuvre des microscopes PALM et est reconnue mondialement pour son expertise dans le développement de la microscopie à très haute résolution pour l’étude de la matière biologique et nanométrique [5].
Références
[1] E. Betzig et al. “Imaging intracellular fluorescent proteins at nanometer resolution” Science 313, 1642–1645 (2006).
[2] F. Diaz et al., “Real-time increase in depth of field of an uncooled thermal camera using several phase-mask technologies”, Opt. Lett. 36 (3), 418-420 (2011).
[3] R. Falcón, F. Goudail, C. Kulcsár, H. Sauer, “Performance limits of binary annular phase masks codesigned for depth-of-field extension,” Opt. Eng. 56(6), 065104 (2017).
[4] O. Lévêque, C. Kulcsár, A. Lee, H. Sauer, A. Aleksanyan, P. Bon, L. Cognet, and F. Goudail, “Co-designed annular binary phase masks for depth-of-field extension in single-molecule localization microscopy”, Opt. Express 28, 32426-32446 (2020).
[5] P. Bon, J. Linarès-Loyez, M. Feyeux, K. Alessandri, B. Lounis, P.Nassoy, L. Cognet, “Self-interference 3D super-resolution microscopy for deep tissue investigations”, Nature Methods 15, 449–454 (2018)